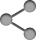임재관 newsmedical@daum.net
오미크론의 전파력을 예측하기 위한 스파이크 단백질의 구조 모델링 분석 결과가 발표됐다.
중앙방역대책본부(본부장 정은경 청장)에 따르면 이번 조사는 오미크론 등 국내 발생 코로나19 변이바이러스 유전자 정보(오미크론, 알파, 델타, 뮤, D614G)를 이용해 바이러스 스파이크 단백질과 감염자의 세포수용체 결합을 분자동역학 모의실험방법으로 분석했다.
그 결과 안정성이 확인됐다.
(그림)코로나19 바이러스(SARS-CoV-2)와 세포수용체 결합 구조
◆오미크론, 결합체 구조 안정성 확인
국내 발생 오미크론, 델타, 알파 등 변이바이러스 스파이크 단백질 유전자 정보를 대상으로 구조적 안정성을 조사한 결과, 오미크론 스파이크 단백질 3개 단위체 간 거리 편차가 가장 낮아 안정적인 구조를 형성했고, 감염자의 세포수용체와 결합자유에너지 분석에서 오미크론이 가장 낮은 에너지 값이 확인돼 결합체 구조 안정성이 확인됐다.
◆바이러스와 세포 간 결합 가능성 증가 확인
이번 분석을 통해, 코로나19 바이러스가 진화하면서 세포와 결합하는 스파이크 단백질 구조의 안정성이 높아졌고, 이에 따라 바이러스와 세포 간 결합 가능성이 증가했음을 확인했다.
이는 발생 초기 코로나19 바이러스와 비교해 세포 결합력 증가에 따른 오미크론 전파력 상승 가능성을 시사한다.
따라서 코로나19 바이러스는 세포 감염 시 구조적 안정성을 높여 결합력을 증가시키는 방향으로 진화하고 있으며, 향후에도 구조적 안정성이 우세한 경향의 변이가 발생할 가능성이 높을 것으로 설명된다.
중앙방역대책본부 정은경 본부장은 “구조 모델링 분석은 충남대학교 강남숙 교수팀과 공동으로 수행됐다”며, “국제 분자 과학 저널(International Journal of Molecular Sciences) 최신호에 게재돼 국내·외 연구진들과 공유될 예정이다”고 밝혔다.
[메디컬월드뉴스]
- TAG



 [정형외과 시술 바로알기]일상생활 속 불편함 유발하는 손목터널증후군·방아쇠수지 각 특징은?
[정형외과 시술 바로알기]일상생활 속 불편함 유발하는 손목터널증후군·방아쇠수지 각 특징은?
 [모발이식 제대로 알기]M자 탈모, 봄에 방치시 여름에 더 빨리 악화…조기 관리 중요
[모발이식 제대로 알기]M자 탈모, 봄에 방치시 여름에 더 빨리 악화…조기 관리 중요
 [치과에 대한 모든 것]눈에 보이지 않는 ‘소아 과잉치’, 정확한 조기 진단이 핵심
[치과에 대한 모든 것]눈에 보이지 않는 ‘소아 과잉치’, 정확한 조기 진단이 핵심
 [피부과 시술 제대로 알기]변형과 통증 유발하는 발톱 무좀, 초기 치료 및 재발 방지 노력 중요
[피부과 시술 제대로 알기]변형과 통증 유발하는 발톱 무좀, 초기 치료 및 재발 방지 노력 중요